DNA phenotyping: Unterschied zwischen den Versionen
Aus exmediawiki
Verena (Diskussion | Beiträge) |
C.heck (Diskussion | Beiträge) Keine Bearbeitungszusammenfassung |
||
| (23 dazwischenliegende Versionen von 2 Benutzern werden nicht angezeigt) | |||
| Zeile 1: | Zeile 1: | ||
='''Workshop und Diskussion zum Thema DNA-Phänotypisierung bzw. erweiterte DNA-Analyse'''= | ='''Workshop und Diskussion zum Thema DNA-Phänotypisierung bzw. erweiterte DNA-Analyse'''= | ||
[[File:Mail DNA.jpg|right|400px|link=https://exmediawiki.khm.de/exmediawiki/images/7/75/Mail_DNA.jpg]] | |||
Mit Matthias Burba (Gast), Dr. Klaus Fritze, Verena Friedrich und Christian Heck | |||
DNA-Phänotypisierung ist die Anwendung von Verfahren, mit denen auf Basis von DNA-Material Rückschlüsse über das äußere Erscheinungsbild eines Individuums gezogen werden können. Diese sogenannte „erweiterte DNA-Analyse” ist in Deutschland noch verboten, eine entsprechende Gesetzesänderung zu deren zukünftiger Anwendung im Strafverfahren ist jedoch in Planung. Mittels „genetischer Phantombilder“ könnten bald Eigenschaften wie das äußere Erscheinungsbild, Alter sowie die bio-geografische Herkunft von Verdächtigten vorhergesagt werden. | |||
In dem dreistündigen Workshop experimentieren und diskutieren wir gemeinsam mit Matthias Burba zu den Möglichkeiten und Grenzen von DNA-Analyseverfahren und deren darauffolgender maschineller Auswertung. | |||
Matthias Burba studierte Jura und Biologie; im Rahmen seiner beruflichen Laufbahn war er u.a. tätig als Justiziar und Leiter der Rechtsabteilung sowie Leiter der Kriminaltechnik der Hamburger Polizei; als Vorsitzender des Unterausschusses Recht und Verwaltung der Innenministerkonferenz sowie als stellvertretender Hamburgischer Datenschutzbeauftragter. | |||
Infomaterial: [https://exmediawiki.khm.de/exmediawiki/images/e/ef/K%C3%B6ln_Reader-DNA.pdf DNA Reader von Matthias Burba] | |||
==classify yourself== | |||
[[Datei:ChelseaManning-ProbablyChelsea.jpg|right|400px]] | |||
''als Eingabedatenset für das in unserem Seminar [https://exmediawiki.khm.de/exmediawiki/index.php/AI@exLabIII Einführung in die Programmierung Künstlicher Intelligenzen] gemeinsam programmierte Perzeptron erstellen wir in diesem Workshop u.a. gemeinsam und experimentell Auszüge aus unserer jeweiligen DNA - unseren genetischen Fingerabdruck.'' | |||
<embedvideo service="youtube">https://www.youtube.com/watch?v=S10l4qS2bcE</embedvideo> | |||
---- | ---- | ||
<big>'''*** WORK-IN-PROGRESS ***'''</big> | |||
==Aktueller Anlass | ==Aktueller Anlass und Kontext== | ||
<br> | <br> | ||
* Was ist DNA-Phänotypisierung? -> die Anwendung von Verfahren, mit denen auf Basis von DNA-Material Rückschlüsse über das äußere Erscheiningsbild eines Individuums gezogen werden | * Was ist DNA-Phänotypisierung? -> die Anwendung von Verfahren, mit denen auf Basis von DNA-Material Rückschlüsse über das äußere Erscheiningsbild eines Individuums gezogen werden | ||
| Zeile 31: | Zeile 47: | ||
* Link zum Projekt: https://deweyhagborg.com/projects/stranger-visions <br> | * Link zum Projekt: https://deweyhagborg.com/projects/stranger-visions <br> | ||
* [https://snapshot.parabon-nanolabs.com/ Parabon NanoLabs], provides DNA phenotyping services for law enforcement organizations. https://en.wikipedia.org/wiki/Parabon_NanoLabs | * [https://snapshot.parabon-nanolabs.com/ Parabon NanoLabs], provides DNA phenotyping services for law enforcement organizations. https://en.wikipedia.org/wiki/Parabon_NanoLabs | ||
* [https://www.genspace.org/ www.genspace.org], NYC community lab (since 2009) | |||
| Zeile 71: | Zeile 88: | ||
---- | ---- | ||
== DNA-Analyse mit dem Bento Lab == | == Praxis-Teil 1: DNA-Analyse mit dem Bento Lab == | ||
<br> | <br> | ||
<embedvideo service="youtube">https://www.youtube.com/watch?v=-Tlq-BTE0FE&feature=youtu.be</embedvideo><br> | <embedvideo service="youtube">https://www.youtube.com/watch?v=-Tlq-BTE0FE&feature=youtu.be</embedvideo><br> | ||
Das Bento Lab ist ein kompaktes DNA-Analyse-Labor. Es beinhaltet: <br> | |||
- Hochgeschwindigkeits-Zentrifuge<br> | |||
- | |||
- PCR-Einheit bzw. Thermocycler<br> | - PCR-Einheit bzw. Thermocycler<br> | ||
- Gelelektrophorese-Kammer<br> | - Gelelektrophorese-Kammer<br> | ||
- Transilluminator<br> | - Transilluminator<br> | ||
<br> | |||
'''Getting started:''' | |||
<br> | |||
* [https://www.bento.bio/protocol/biotechnology-101/ Biotechnology 101], eine Einführung zur Benutzung des Bento Labs in Kombination mit dem mitgelieferten Starter Kit. | |||
<br> | |||
'''Experiment:<br>''' | |||
* auf Basis unterschiedlicher Speichelproben das Gen ACTN3 analysieren und visualisieren, das mit sportlicher Performance assoziiert wird. | |||
* https://www.bento.bio/protocol/biotechnology-101/athlete-gene/ | |||
* https://de.wikipedia.org/wiki/Alpha-Actinin_3 | |||
* https://www.wissenschaft.de/umwelt-natur/entscheidet-ein-gen-zwischen-sprinter-und-ausdauersportler/ | |||
* https://futurezone.at/science/der-dna-analyseboom-im-web/24.565.172 | |||
---- | |||
==Praxis-Teil 2: Genotype-Phenotype Mapping== | |||
== | |||
<br> | <br> | ||
'''Genetic information test data (23andMe) as used by Heather for her first tests''' | '''Genetic information test data (23andMe) as used by Heather for her first tests:''' | ||
<br> | <br> | ||
* [https://github.com/orta/dna DNA as raw text by user Orta], released under Creative Commons Attribution on Github | * [https://github.com/orta/dna DNA as raw text by user Orta], released under Creative Commons Attribution on Github | ||
* [https://github.com/msporny/dna Manu Sporny's genetic information], released under Creative Commons Public Domain License (CC0) on Github | * [https://github.com/msporny/dna Manu Sporny's genetic information], released under Creative Commons Public Domain License (CC0) on Github | ||
<br> | |||
'''Public version of the genetic profiling code by Heather-Dewey-Hagborg:'''<br> | |||
* https://github.com/hdeweyh/strangerVisions | |||
<br> | |||
'''Python requirements<br>''' | |||
* python==2.7 | |||
* biopython==1.68 | |||
* CherryPy==8.1.2 | |||
* Jinja2==2.8 | |||
* MarkupSafe==0.23 | |||
* six==1.10.0 | |||
* texttable==0.8.7 | |||
---- | |||
<!-- | |||
== Next: DNA-Sequenzierung mit dem MinIon Sequencer== | |||
<br> | |||
* https://nanoporetech.com/products/minion | |||
* https://www.foodrepo.org/ch/dna & http://wiki.hackuarium.ch/w/Open-Food-DNA?fbclid=IwAR0u7t1BATjXbfxokXygxSs7hTPTQfuw08JZXTnrPxcRyQrGXgg1YcA6ZHk | |||
---- | |||
--> | |||
Aktuelle Version vom 23. März 2021, 11:18 Uhr
Workshop und Diskussion zum Thema DNA-Phänotypisierung bzw. erweiterte DNA-Analyse

Mit Matthias Burba (Gast), Dr. Klaus Fritze, Verena Friedrich und Christian Heck
DNA-Phänotypisierung ist die Anwendung von Verfahren, mit denen auf Basis von DNA-Material Rückschlüsse über das äußere Erscheinungsbild eines Individuums gezogen werden können. Diese sogenannte „erweiterte DNA-Analyse” ist in Deutschland noch verboten, eine entsprechende Gesetzesänderung zu deren zukünftiger Anwendung im Strafverfahren ist jedoch in Planung. Mittels „genetischer Phantombilder“ könnten bald Eigenschaften wie das äußere Erscheinungsbild, Alter sowie die bio-geografische Herkunft von Verdächtigten vorhergesagt werden.
In dem dreistündigen Workshop experimentieren und diskutieren wir gemeinsam mit Matthias Burba zu den Möglichkeiten und Grenzen von DNA-Analyseverfahren und deren darauffolgender maschineller Auswertung.
Matthias Burba studierte Jura und Biologie; im Rahmen seiner beruflichen Laufbahn war er u.a. tätig als Justiziar und Leiter der Rechtsabteilung sowie Leiter der Kriminaltechnik der Hamburger Polizei; als Vorsitzender des Unterausschusses Recht und Verwaltung der Innenministerkonferenz sowie als stellvertretender Hamburgischer Datenschutzbeauftragter.
Infomaterial: DNA Reader von Matthias Burba
classify yourself

als Eingabedatenset für das in unserem Seminar Einführung in die Programmierung Künstlicher Intelligenzen gemeinsam programmierte Perzeptron erstellen wir in diesem Workshop u.a. gemeinsam und experimentell Auszüge aus unserer jeweiligen DNA - unseren genetischen Fingerabdruck.
*** WORK-IN-PROGRESS ***
Aktueller Anlass und Kontext
- Was ist DNA-Phänotypisierung? -> die Anwendung von Verfahren, mit denen auf Basis von DNA-Material Rückschlüsse über das äußere Erscheiningsbild eines Individuums gezogen werden
- diese DNA-Phänotypisierung bzw. "erweiterte DNA-Analyse" ist in Deutschland auf Grund der rechtlichen Beschränkungen in der Strafprozessordnung bisher verboten
- laut Koalitionsvertrag der aktuellen Bundesregierung von 2018 ist geplant, die herkömmliche/forensische DNA-Analyse im Strafverfahren im Sinne der erweiterten DNA-Analyse auszuweiten, und zwar auf Merkmale wie Augenfarbe, Haarfarbe, Hautfarbe sowie Alter
- Bisher durften nur die nichtkodierenden Abschnitte der DNA analysiert werden, nicht aber die codierenden Abschnitte (auf denen genau diese in der erweiterten DNA-Analyse untersuchten Merkmale liegen).
- auf den kodierenden Abschnitten liegen auch Informationen zu Krankheiten etc., deren Erfassung man in der erweiterten DNA-Analyse zwar nicht anstrebt, aber auch nicht ausschließen kann – insofern könnte die EDA Tür und Tor zur Erfassung solch präkerer Informationen öffnen
- der Einsatz dieser neuen Methode ist unter gewissen Voraussetzungen in den Niederlanden, Frankreich, Spanien und den USA bereits erlaubt.
- das Gesetz ist in erster Instanz schon entschieden, und wird wohl kommen...
Links:
- www.wikipedia.org/DNA-Phänotypisierung
- Artikel: Mit DNA ein Phantombild zeichnen
- Artikel: Noch kein biologisches Phantombild möglich
- Stellungnahme der Spurenkommission zu den “Möglichkeiten und Grenzen der DNA-gestützten Vorhersage äußerer Körpermerkmale, der biogeographischen Herkunft und des Alters unbekannter Personen anhand von Tatortspuren im Rahmen polizeilicher Ermittlungen” (2016)
Beispiel: Kunstprojekt "Stranger Visions"
"In Stranger Visions I collected hairs, chewed up gum, and cigarette butts from the streets, public bathrooms and waiting rooms of New York City. I extracted DNA from them and analyzed it to computationally generate 3d printed life size full color portraits representing what those individuals might look like, based on genomic research. Working with the traces strangers unwittingly left behind, the project was meant to call attention to the developing technology of forensic DNA phenotyping, the potential for a culture of biological surveillance, and the impulse towards genetic determinism.
The forecast of Stranger Visions came true. Just 2 years later Parabon NanoLabs launched a service they called DNA "snapshot" to police around the US. For more examples see Identitas and read about their collaboration with the Toronto police."
- Link zum Projekt: https://deweyhagborg.com/projects/stranger-visions
- Parabon NanoLabs, provides DNA phenotyping services for law enforcement organizations. https://en.wikipedia.org/wiki/Parabon_NanoLabs
- www.genspace.org, NYC community lab (since 2009)
DNA fingerprinting vs. DNA phenotyping
Der bisherige Gesetzestext (§ 81e Molekulargenetische Untersuchung) soll mit folgendem Satz ergänzt werden: "Ist unbekannt, von welcher Person das Spurenmaterial stammt, dürfen zusätzlich Feststellungen über das Geschlecht, die Augen-, Haar-und Hautfarbe sowie das biologische Alter der Person getroffen werden."
DNA-Phänotyping bzw. EDA soll nämlich in Zukunft vor allem eingesetzt werden, wenn der/die Täter/in gänzlich unbekannt ist. Auf dieser Basis könnte dann ein genetisches/biologisches Phantombild entstehen, welches man zur Tätersuche einsetzen bzw. wodurch man die Menge der Verdächtigen eingrenzen kann.
Von dieser Gruppe der Verdächtigen würden dann DNA-Proben genommen (z.B. Mundabstrich), die dann gegen eine vorhandene Referenz-DNA des/der Täter/in abgeglichen werden kann. Diesen Schritt bezeichnet man als DNA-Fingerprinting. Bei der herkömmlichen DNA-Analyse gleicht man die Ergebnisse mit Einträgen in einer Datenbank ab, z.B. der des Bundeskriminalamts, oder vergleicht mit einer Referenz-DNA.
Wiki-Artikel DNA-Fingerprinting:
"Für den genetischen Fingerabdruck werden derzeit zwischen 8 und 15 Abschnitte aus der DNA mit Hilfe der PCR-Methode vervielfältigt. Bei dieser Methode werden nur nicht-codierende Bereiche der DNA untersucht."
"Im Gegensatz zu anderen DNA-Analysen, bei denen mittels Sequenzierungen Gene aus den codierenden Bereichen der DNA untersucht werden, die durchaus Rückschlüsse etwa auf eventuelle Krankheiten des Individuums zulassen, lassen sich aus dem Zahlencode der Fragmentlängen-Analyse so gut wie keine Eigenschaften des Individuums ableiten.[4] Über einen zusätzlichen Locus wird allerdings das Geschlecht bestimmt. Bestimmte Abweichungen in der Anzahl der Chromosomen, wie die dem Down-Syndrom zugrundeliegenden, werden ebenfalls offenbart."
PDF Genetischer Fingerabdruck und DNA-Analyse: Was steckt dahinter?
Beispiel: DNA-Fingerprinting
Familienuntersuchungen
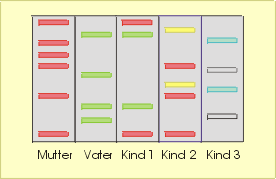
Die Abbildung demonstriert an einem fiktiven Beispiel das Ergebnis der Untersuchung einer Familie bestehend aus Vater, Mutter, einem gemeinsamen Kind von Vater und Mutter (Kind 1), einem Kind der Mutter aus einer vorausgegangenen Ehe (Kind 2) und einem adoptierten Kind (Kind 3). (Die einzelnen Banden wurden verschiedenfarbig dargestellt, um die familiären Zusammenhänge zu verdeutlichen und entsprechen nicht den Farbmarkierungen der zur Untersuchung benutzten Primer)
Quelle: https://www.meine-molekuele.de/der-genetische-fingerabdruck/
Praxis-Teil 1: DNA-Analyse mit dem Bento Lab
Das Bento Lab ist ein kompaktes DNA-Analyse-Labor. Es beinhaltet:
- Hochgeschwindigkeits-Zentrifuge
- PCR-Einheit bzw. Thermocycler
- Gelelektrophorese-Kammer
- Transilluminator
Getting started:
- Biotechnology 101, eine Einführung zur Benutzung des Bento Labs in Kombination mit dem mitgelieferten Starter Kit.
Experiment:
- auf Basis unterschiedlicher Speichelproben das Gen ACTN3 analysieren und visualisieren, das mit sportlicher Performance assoziiert wird.
- https://www.bento.bio/protocol/biotechnology-101/athlete-gene/
- https://de.wikipedia.org/wiki/Alpha-Actinin_3
- https://www.wissenschaft.de/umwelt-natur/entscheidet-ein-gen-zwischen-sprinter-und-ausdauersportler/
- https://futurezone.at/science/der-dna-analyseboom-im-web/24.565.172
Praxis-Teil 2: Genotype-Phenotype Mapping
Genetic information test data (23andMe) as used by Heather for her first tests:
- DNA as raw text by user Orta, released under Creative Commons Attribution on Github
- Manu Sporny's genetic information, released under Creative Commons Public Domain License (CC0) on Github
Public version of the genetic profiling code by Heather-Dewey-Hagborg:
Python requirements
- python==2.7
- biopython==1.68
- CherryPy==8.1.2
- Jinja2==2.8
- MarkupSafe==0.23
- six==1.10.0
- texttable==0.8.7